Manlio Migliorati – Università degli Studi di Brescia
Maurizio Carpita – Università degli Studi di Brescia
L’emergenza sanitaria mondiale causata in questi primi mesi del 2020 dal nuovo coronavirus COVID-19 rende ancor più importante la disponibilità di informazioni scientifiche affidabili, in primo luogo per favorire i contatti tra gruppi di ricerca che si occupano dello stesso tema ma anche per contrastare la diffusione di fake news. In uno scenario ancora di piena epidemia, uno dei problemi più rilevanti è quello di far circolare in tempi brevi la conoscenza sulle ricerche in corso, così da permettere a chi da varie prospettive affronta complesse criticità sanitarie, economiche e sociali di prendere decisioni il più possibile consapevoli e di conoscere le possibili soluzioni già sperimentate da altri.
Al DMS StatLab dell’Università di Brescia analizziamo varie banche dati internazionali che contengono informazioni sulle pubblicazioni scientifiche che riguardano il nuovo coronavirus, da quelle più note e “istituzionali”, come Web of Science (WoS) e SCOPUS, a quelle create proprio in occasione di questa emergenza per favorire la open research, come quella della World Health Organization [1] o come CORD-19, archivio reso disponibile dall’Allen Istitute for Artificial Intelligence [2] che al 27 marzo 2020 contiene più di 45 mila pubblicazioni scientifiche sull’argomento, di cui 33 mila articoli in full text.
In questo breve articolo presentiamo un semplice esempio di come l’applicazione di tecniche statistiche di analisi e visualizzazione dei dati utilizzabili per il cosiddetto “text mining” offra l’opportunità di individuare i temi più rilevanti correlati con l’emergenza causata da COVID-19, le principali fonti informative e le istituzioni dei paesi che si stanno impegnando in queste ricerche.
Per questa analisi abbiamo considerato le 343 pubblicazioni scientifiche (articoli, lettere, news e altro materiale editoriale indicizzato) selezionati da WoS per il periodo dal 1 gennaio al 30 marzo 2020 utilizzando le seguenti 11 parole chiave (senza distintinzione tra lettere maiuscole e minuscole): COVID-19, COVID2019, COVID-2019, COVID 2019, COV19, 2019-nCoV, SARS-CoV-2, SARS-Cov2, coronavirus disease 2019, Novel Coronavirus, New Coronavirus.
Le 343 pubblicazioni sono state prima di tutto classificate con la procedura topic modeling [3], molto utilizzata per il natural language processing e il machine learning, che permette di individuare i temi trattati nei documenti disponibili. I risultati ottenuti nel nostro caso considerando titolo, abstract e keywords hanno suggerito di suddividere le pubblicazioni in tre gruppi (l’algoritmo non è stato in grado di classificare con sufficiente certezza 2 pubblicazioni). La successiva lettura delle informazioni disponibili per i primi 5 top articles di ogni gruppo ha permesso di inviduare in prima approssimazione tre macro-temi: “diffusione e impatto sociale di COVID-19″ (100 pubblicazioni), “relazioni tra COVID-19 e altri virus già conosciuti” (109 pubblicazioni) e “diagnosi, trasmissione e caratteristiche cliniche di COVID-19″ (132 pubblicazioni).
Successivamente i tre gruppi di pubblicazioni sono stati analizzati utilizzando le tecniche di visualizzazione messe a disposizione dalla applicazione bibliometrix [4]. Per brevità e a titolo di esempio mostriamo solo alcuni risultati ottenuti per il terzo gruppo di pubblicazioni, che risulta composto da 40 articoli, 27 lettere, 14 news e con 51 altri materiali editoriali indicizzati.
La Co-occurence Network (Fig. 1 a sinistra) rappresenta la rete delle relazioni tra 4 gruppi di parole chiave, due chiaramente riferiti ai vari nomi di COVID-19, uno riferito alla gestione del rischio a Wuhan e uno che rimane isolato riferito alla diagnosi e al trattamento. La Collaboration Network (Fig. 1 a destra) rappresenta la rete delle relazioni tra i paesi dei gruppi di ricerca autori delle pubblicazioni: sono individuati 3 gruppi di paesi, il primo collegato alla Cina (ma con la presenza inattesa della Spagna), il secondo collegato agli Stati Uniti e all’Europa (con la presenza inattesa dell’Arabia Saudita), il terzo isolato e composto da soli due paesi (Germania e Svezia). L’applicazione correda questi grafici con tabelle che mostrano per ogni paese della rete il numero di pubblicazioni associate ed è successivamente possibile filtrare ed estrarre quelle di interesse.
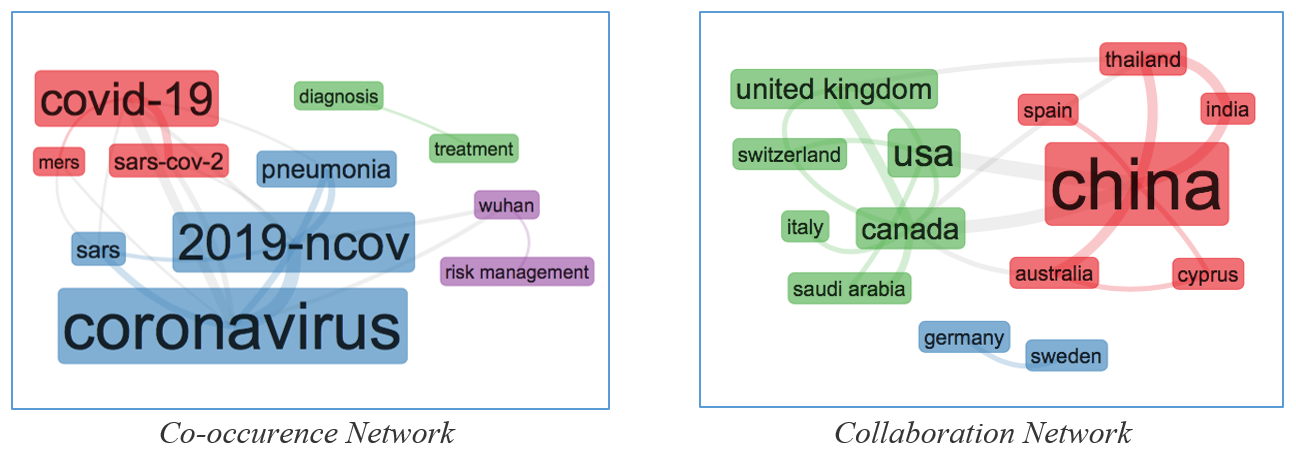
Fig. 1 Visualizzazione delle relazioni tra parole chiave (a sinistra) e tra paesi dei gruppi di ricerca (a destra) di 132 pubblicazioni su diagnosi, trasmissione e caratteristiche cliniche di COVID-19 (WoS 30.03.2020)
Il Three-Field Plot (Fig. 2) rappresenta invece i collegamenti tra parole chiave più importanti (a sinistra), istituzioni di ricerca (al centro) e collocazione editoriale (a destra) delle 132 pubblicazioni che riguardano diagnosi, trasmissione e caratteristiche cliniche di COVID-19 (per favorirne la leggibilità, da questo grafico abbiamo eliminato le parole COVID-19 e i suoi sinonimi).
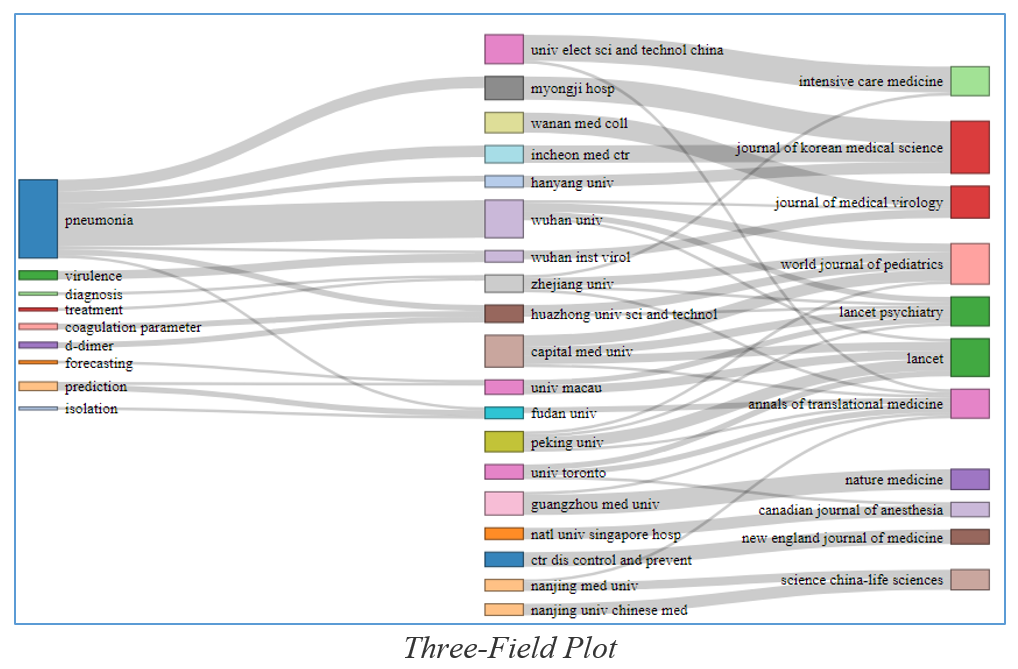
Fig. 2 Visualizzazione delle relazioni tra parole chiave, istituzioni di ricerca e collocazione editoriale di 132 pubblicazioni su diagnosi, trasmissione e caratteristiche cliniche di COVID-19 (WoS 30.03.2020)
Oltre a parole chiave generiche come “pneumonia” (ovvero “polmonite”, che caratterizza comunque solo un quinto delle pubblicazioni), emergono anche termini come “d-dimer” e “coagulation parameter” che possono essere di particolare interesse per i ricercatori, i quali muovendosi con il mouse sui rettangoli colorati del grafico possono evidenziare il numero di pubblicazioni collegate e anche in questo caso possono successivamente filtrare ed estrarre quelle di specifico interesse. Il Three-Field Plot si può facilmente modificare sostituendo le istituzioni con i nomi degli autori principali (in pratica i referenti dei gruppi di ricerca), così da collegare le parole chiave dei loro studi con la collocazione editoriale dei risultati che hanno ottenuto.
Il numero di pubblicazioni scientifiche su COVID-19 presenti in WoS che si possono analizzare e visualizzare con tecniche statistiche come quelle qui mostrate cresce velocemente di giorno in giorno e ulteriori possibilità ancora poco esplorate sono offerte dall’integrazione con altre banche dati orientate alla open research come PubMed, arXiv, bioRxiv e medRxiv [5]. Una più grande disponibilità ma anche una migliore qualità di questi archivi, nonché un maggior utilizzo delle tecnologie di analisi e visualizzazione di questi dati può oggi favorire la diffusione delle informazioni e della conoscenza scientifica in tempi molto più brevi e con modalità molto più efficaci di quanto accadeva in passato.
Sitografia
[2] pages.semanticscholar.org/coronavirus-research
[3] www.tidytextmining.com/topicmodeling.html
[5] www.ncbi.nlm.nih.gov/pubmed/ ; arxiv.org ; www.biorxiv.org ; www.medrxiv.org

